肺炎球菌の進化過程で保存された病原因子の探索
担当: 山口雅也
ひとつの菌種が存続していく過程において、宿主の抗体や抗菌薬などの選択が働いた場合、結合部位の変異といった正の選択が働くことで多様性が生じる。一方で、自然免疫機構の回避など、菌の生存に重要な役割を果たす分子には、機能を維持するため負の選択が生じると考えられる。本研究では、Streptococcus pneumoniaeの特徴的な分子であるコリン結合タンパク質群について、種内における系統関係と選択圧の解析を行った。
全ゲノムが解読されたS. pneumoniae 28株について、BLAST検索により16種類のコリン結合タンパク質を選出した。各コリン結合タンパク質をコードする遺伝子群について、ベイズ法による分子系統樹をそれぞれ作成し、オルソログの関係にある遺伝子を判定した。さらに、得られたオルソロガスな遺伝子配列群と系統樹から、ベイズ推定に基づく分子進化解析を行った。その結果、16種類のコリン結合タンパク質群のうち、cbpJとlytAの2種類の遺伝子のみにおいて、10%以上のコドンが負の選択下にあることが示された。cbpC, cbpG, cbpLの3遺伝子は、各菌株における配列で共通する相同なコドン領域を持たなかった。次に、S. pneumoniae TIGR4株を親株として、cbpJ, cbpL遺伝子欠失株をそれぞれ作製した。各菌株を用いて好中球殺菌試験を行ったところ、感染3時間後に、TIGR4野生株と比較して、cbpJ欠失株の生存率が有意に低下した。また、マウス経鼻感染モデルにおいて、野生株感染群と比較して、cbpJ欠失株感染群の死亡率が有意に低かった。一方で、cbpL欠失株では、野生株と比較して病原性が低下する傾向が認められたが、有意な差は示さなかった。
以上の結果から、S. pneumoniaeのコリン結合タンパク質群において、CbpJとLytAがとくに強い負の制御下にあることが示された。さらに、CbpJはS. pneumoniaeの肺感染時の病原因子として働くことが示唆された。
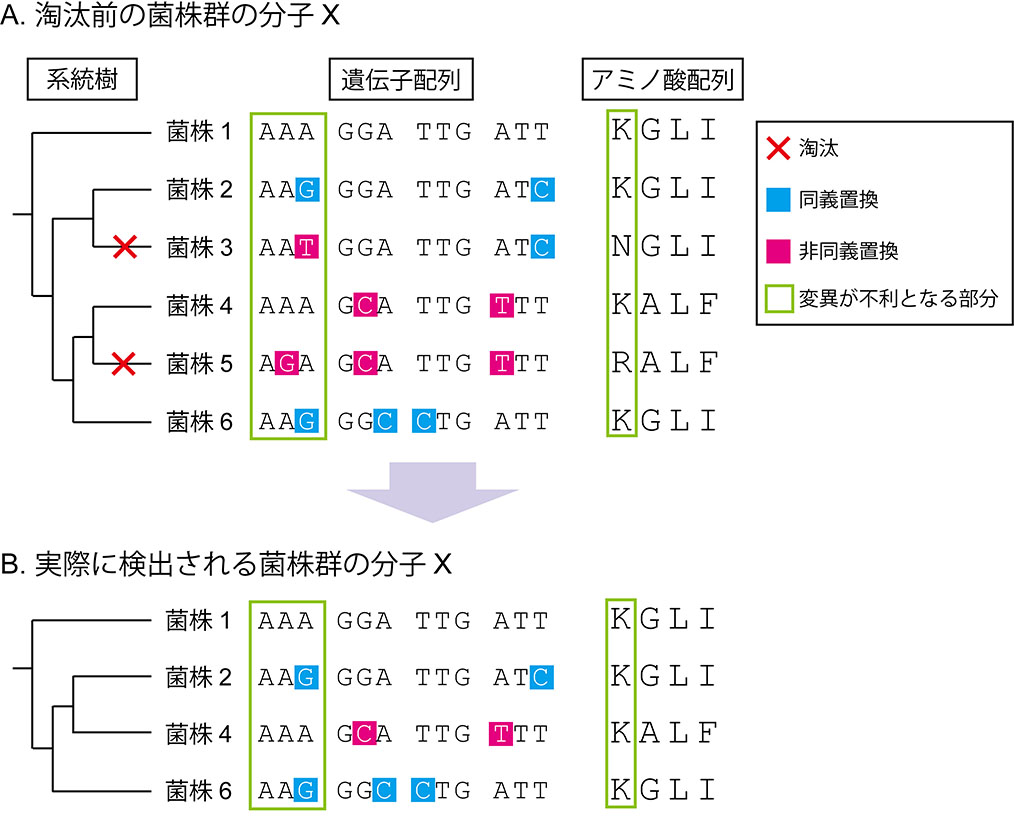
図. 分子進化解析の概念
ランダムに遺伝子変異が起こると、同義置換と非同義置換はほぼ等確率に発生する (A)。しかし実際は、その菌の生存を妨げるような変異は淘汰される (B)。その結果、進化の過程において、菌の生存に重要な役割を果たす遺伝子には同義置換が蓄積されていくと考えられる。本研究計画では、ゲノム情報をもとに統計学的な解析を行い、同義置換が蓄積されている分子を選出する。さらに選出された分子について、病原性に果たす役割とワクチン抗原としての機能を評価する。
関連文献
Yamaguchi M., Goto K., Hirose Y., Yamaguchi Y., Sumitomo T., Nakata M., Nakano K., Kawabata S. Identification of evolutionarily conserved virulence factor by selective pressure analysis of Streptococcus pneumoniae. Commun. Biol., Vol. 2. 96. 2019.
Yamaguchi M., Synergistic findings from microbiological and evolutional analyses of virulence factors among pathogenic streptococcal species. J. Oral Biosci., Vol. 60. pp36-40. 2018. invited Review.
Yamaguchi M., Nakata M., Sumioka R., Hirose Y., Wada S., Akeda Y., Sumitomo T., Kawabata S. Zinc metalloproteinase ZmpC suppresses experimental pneumococcal meningitis by inhibiting bacterial invasion of central nervous systems. Virulence, Vol. 8. pp1516-1524. 2017.
Yamaguchi M., Hirose Y., Nakata M., Uchiyama S., Yamaguchi Y., Goto K., Sumitomo T., Lewis A.L., Kawabata S., Nizet V. Evolutionary inactivation of a sialidase in group B Streptococcus. Sci. Rep., Vol. 6. 28852. 2016.